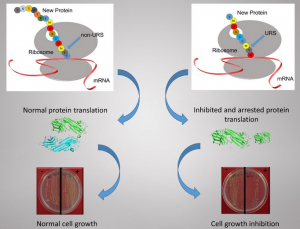
Le Pr Noam Adir du Technion a voulu mettre à profit l’accès aux séquences des génomes connus pour une recherche originale, celle des séquences absentes. Y a-t-il des séquences courtes sous-représentées par rapport à l’abondance à laquelle on s’attendrait à les trouver si leur distribution était aléatoire ? Le Pr Adir et son équipe, en particulier Dr. Sharon Penias-Navon et Tali Schwartzman, se sont alors mis à la recherche des séquences perdues qu’ils ont appelées URS pour under-representated sequences. Il s’agit de séquences d’ADN d’une douzaine de nucléotides qui sont très peu abondantes voire absentes d’un organisme donné. Le premier organisme étudié par les chercheurs a été la bactérie Escherichia coli. Ils ont observé que certaines séquences courtes d’ADN ne peuvent être lues par la machinerie cellulaire de cette bactérie pour être traduites en protéines. Quand un ribosome rencontre ces séquences, il s’arrête et la traduction est stoppée, ce qui affecte la croissance des bactéries.
Les URS sont spécifiques à une espèce donnée : celles qui interfèrent avec les ribosomes d’Escherichia coli laissent de marbre les nôtres. En fait, les nôtres ne semblent être arrêtés par rien, les chercheurs n’ayant pas trouvé d’URS humains. Ceci est un argument de plus en faveur d’une utilisation potentielle de ces URS comme antibiotiques, comme les chercheurs le suggèrent. Un brevet a déjà été déposé dans ce sens.
Publication scientifique dans PNAS, juin 2016
Source bvst, par Tirtsa Ackermann, doctorante, Université Hébraïque de Jérusalem